Stage LPS - Détection automatique de flakes 2D
Stage de 2 mois au LPS : détection automatique de flakes (graphène, hBN) et prédiction des couches.
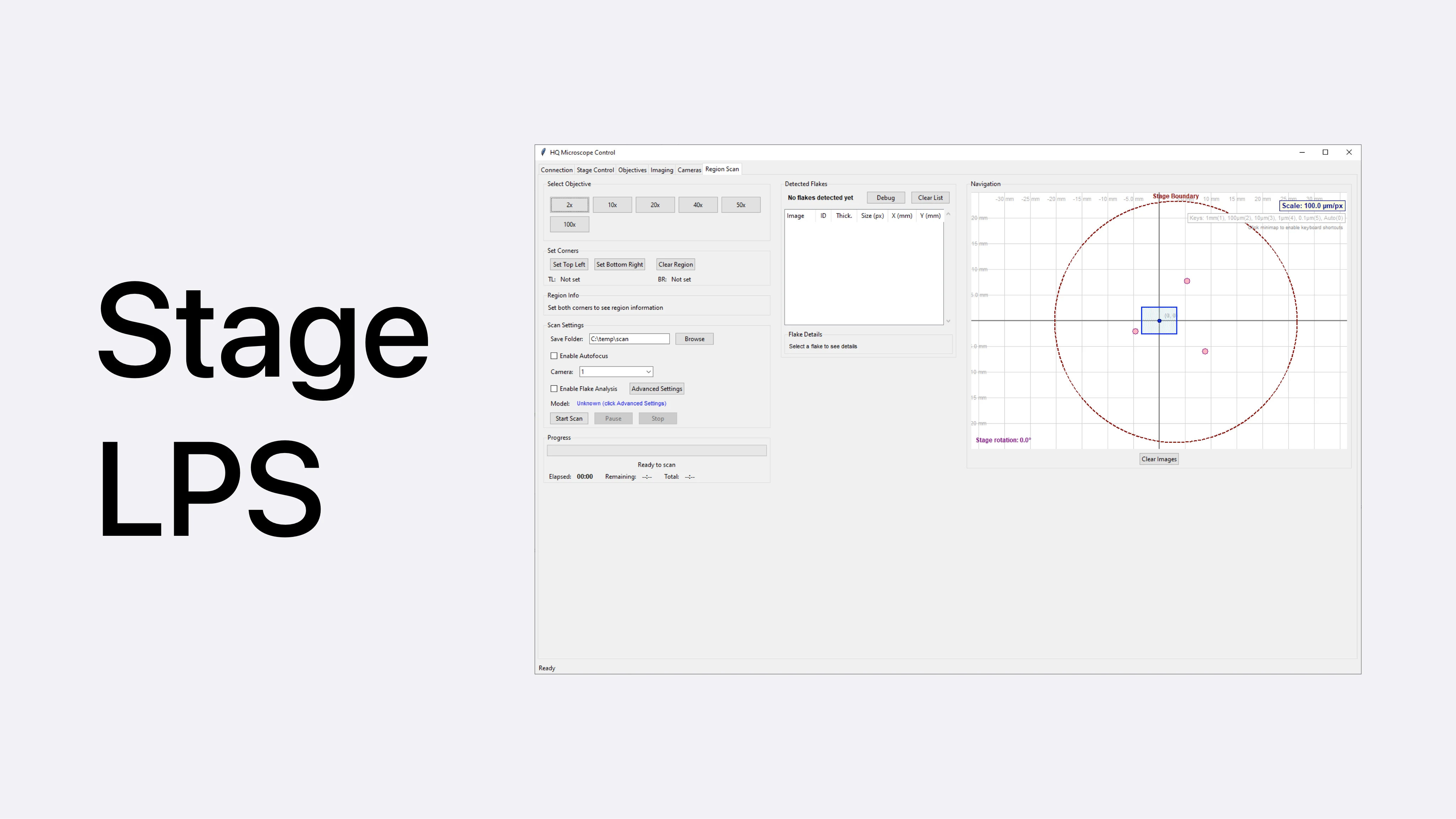
Capture d'écran du logiciel développé
Technologies utilisées :
- IA / Vision : Python, segmentation + classification, approche avec MaskTerial
- Données : acquisition terrain, annotation LabelMe, calibration AFM
- Instrumentation : microscope HQ Graphene motorisé (XY, autofocus, caméra), AFM
- Application : PyQt (scan automatique, visualisation, export, reprise de scan)
Résumé du projet
Dans le cadre de mon stage de fin de première année au LPS (CNRS / Université Paris-Saclay), j’ai développé en 2 mois un pipeline complet de détection automatique de flakes 2D (graphène, hBN), de la création du jeu de données à l’application finale.
Problème de départ
Au laboratoire, la recherche de flakes sur wafer est une tâche lente et manuelle : balayage XY sur de grandes zones, vérification visuelle, puis retour sur les candidats pour caractérisation.
Objectif du stage : remplacer cette phase par un flux automatisé, exploitable par les équipes.
Fonctionnalités principales
- Scan automatique d’un échantillon (balayage XY + autofocus + capture)
- Localisation automatique des flakes
- Prédiction du nombre de couches
- Estimation de la taille et de la forme des flakes
- Visualisation avec minimap + superposition des images acquises en direct
- Export des résultats
- Reprise d’un scan interrompu ou chargement d’un scan précédent
Backbone scientifique et technique
1) Acquisition + annotation
- Constitution d’un jeu de données interne en scannant des échantillons
- Annotation d’environ 100 flakes avec LabelMe (polygones + classes)
2) Modélisation IA (récit final retenu)
- Choix d’une approche MaskTerial (modèle de fondation + classifieur adaptable)
- Utilisation de poids pré-entraînés (contrainte de calcul), puis adaptation aux données locales
- Pipeline cible plus flexible qu’un modèle rigide par matériau/microscope
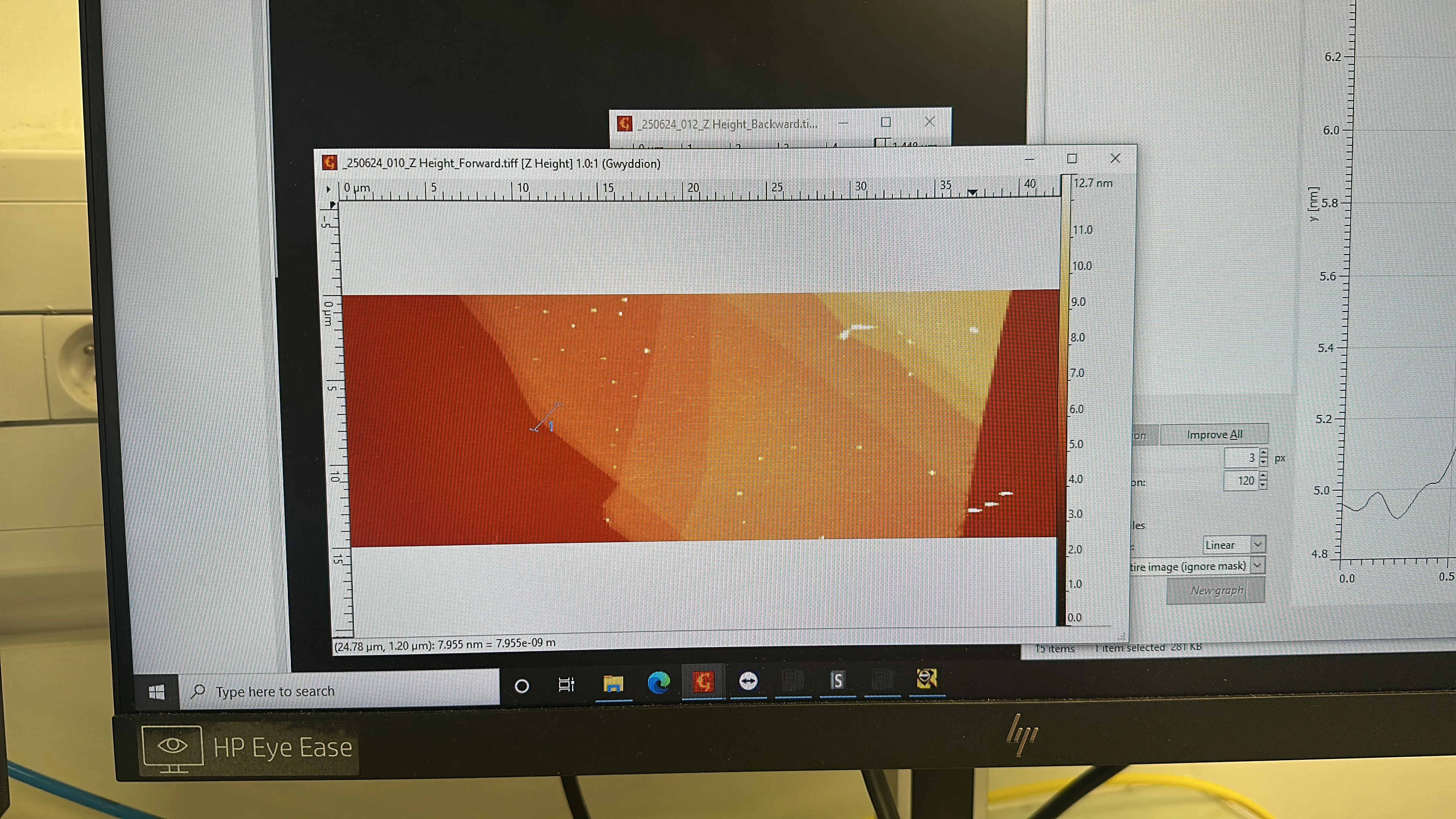
3) Calibration AFM (nombre de couches)
- Comptage basé sur le pas entre couches de graphène
- Référence silicium/graphène calibrée à partir d’un échantillon connu (base = 1 couche)
4) Intégration instrumentale
- Application PyQt interfaçant le microscope HQ Graphene via le logiciel fournisseur + pipe
- Exploration d’un second microscope sans API officielle : reverse engineering des DLL pour récupérer les contrôles XY/autofocus/caméra
Défis techniques rencontrés
- Traiter des objets minuscules et des classes rares (entraînement/prédiction instables)
- Éviter une approche trop rigide et difficile à maintenir
- Intégrer proprement IA + acquisition microscope dans un workflow unique
- Garantir des performances suffisantes pour une utilisation en conditions réelles
- Maintenir la qualité malgré la sensibilité au couple microscope/caméra
Résultats et impact
- Inférence d’environ 0,5-0,6 s/image sur GTX 1080 Ti (plus rapide que la vitesse de scan)
- Gain de plusieurs heures par rapport à une recherche manuelle
- Détection de flakes qui auraient pu être manqués visuellement
- Traçabilité améliorée (images + prédictions + métadonnées)
- Base logicielle réutilisable pour d’autres matériaux (avec jeu de données adapté)
Ce que j’ai appris
Ce projet m’a permis de :
- Construire un système complet de computer vision pour un usage en laboratoire
- Relier des mesures expérimentales (AFM) et des modèles IA pour une prédiction exploitable
- Concevoir une application Python robuste autour d’un instrument motorisé
- Livrer un outil utile en autonomie complète, de la recherche à la mise en service